Trabajo desde consola con YASARA.
Quiero ejecutar un macro de YASARA que abra un fichero YOB, seleccione una molécula con el ligando UNL 1, cambie el nombre de la cadana a B y guarde la estructura modificada con el nombre que desee. ¿Cómo automatizar este proceso para repetirlo 476 veces...?
Empiezo por crear un fichero "_template-YASARA-cambio-UNL1-to-B.txt" que usaré como plantilla y que será el que proporcione las órdenes que debe ejecutar YASARA.
# Copia y pega el contenido de este script en un fichero
# de texto llamado "_template-YASARA-cambio-UNL1-to-B.txt"
# Modifica las rutas donde se localizan los ficheros de acuerdo a tus necesidades.
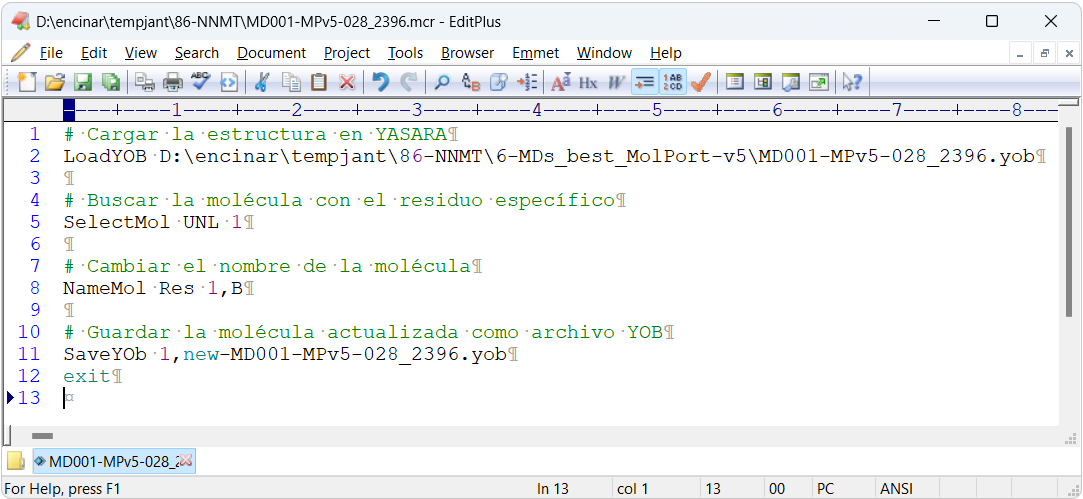
# Cargar la estructura en YASARA
LoadYOB D:\encinar\tempjant\86-NNMT\6-MDs_best_MolPort-v5\[variable-1].yob
# Selecciona la molécula con el residuo específico: UNL 1
SelectMol UNL 1
# Cambiar el nombre de la molécula
NameMol Res 1,B
# Guardar la molécula actualizada como archivo YOB, con nombres de tipo [variable-2].yob
SaveYOb 1,[variable-2].yob
exitGenera otro fichero de texto llamado _dos_columnas.txt que contenga dos variables en dos columnas, separadas por tabuladores. La primera columna contiene los nombres de cada fichero YOB (en mi caso, cada fichero YOB contiene la estructura de una proteína con una molécula orgánica acoplada en su sitio catalítico, ambas moléculas tienen por nombre A y queremos que la molécula orgánica cambie a B). La segunda columna tiene los nombres que queremos que tengan los nuevos ficheros generados, he añadido el prefijo "new-" al nombre de cada fichero YOB ya existente.
MD001-MPv5-028_2396 new-MD001-MPv5-028_2396
MD002-MPv5-155_1658 new-MD002-MPv5-155_1658
MD003-MPv5-139_2600 new-MD003-MPv5-139_2600
MD004-MPv5-146_220 new-MD004-MPv5-146_220
MD005-MPv5-035_212 new-MD005-MPv5-035_212
MD006-MPv5-137_3838 new-MD006-MPv5-137_3838
MD007-MPv5-137_3856 new-MD007-MPv5-137_3856
MD008-MPv5-139_2579 new-MD008-MPv5-139_2579
MD009-MPv5-143_1968 new-MD009-MPv5-143_1968
MD010-MPv5-151_849 new-MD010-MPv5-151_849
MD011-MPv5-137_3840 new-MD011-MPv5-137_3840Escribiremos el siguiente script de Python en un fichero llamado YASARA-mcr-cambio-RES-UNL-1-B.py, este script se ejecuta de forma muy cómodo desde Visual Studio code, como un archivo de Jupyter Notebook. La ejecución de este script generará 11 ficheros con extensión *.mcr y cada uno de los nombres de la [variable-1].
import os
# Rutas de los archivos
template_file_path = r"D:\6-MDs_best_MolPort-v5\_template-YASARA-cambio-UNL1-to-B.txt"
var_file_path = r"D:\6-MDs_best_MolPort-v5\_dos_columnas.txt"
output_dir = r"D:\6-MDs_best_MolPort-v5"
# Leer el archivo de plantilla
with open(template_file_path, 'r', encoding='iso-8859-1') as template_file:
template = template_file.read()
# Leer el archivo de variables y procesar cada línea
with open(var_file_path, 'r', encoding='iso-8859-1') as var_file:
for line in var_file:
# Separar las variables por tabuladores y eliminar espacios en blanco
variables = [var.strip() for var in line.split('\t')]
# Obtener los valores de las variables
variable_1 = variables[0]
variable_2 = variables[1]
# Generar el contenido de salida reemplazando las variables en la plantilla
output_content = template.replace('[variable-1]', variable_1).replace('[variable-2]', variable_2)
# Construir la ruta de salida y guardar el archivo
output_file_name = f"{variable_1}.mcr"
output_file_path = os.path.join(output_dir, output_file_name)
with open(output_file_path, 'w', encoding='iso-8859-1') as output_file:
output_file.write(output_content)
print("Proceso completado con éxito!")Como ejemplo, mira justo debajo el contenido del fichero MD001-MPv5-028_2396.mcr:
Desde la consola CMD puedes pedir a YASARA que ejecute este macro con el siguiente código (sin las explicaciones marcadas por #):
# YASARA llamará al macro en la ruta deseada y este actuará sobre el fichero MD001-MPv5-028_2396, localizado en la ruta deseada
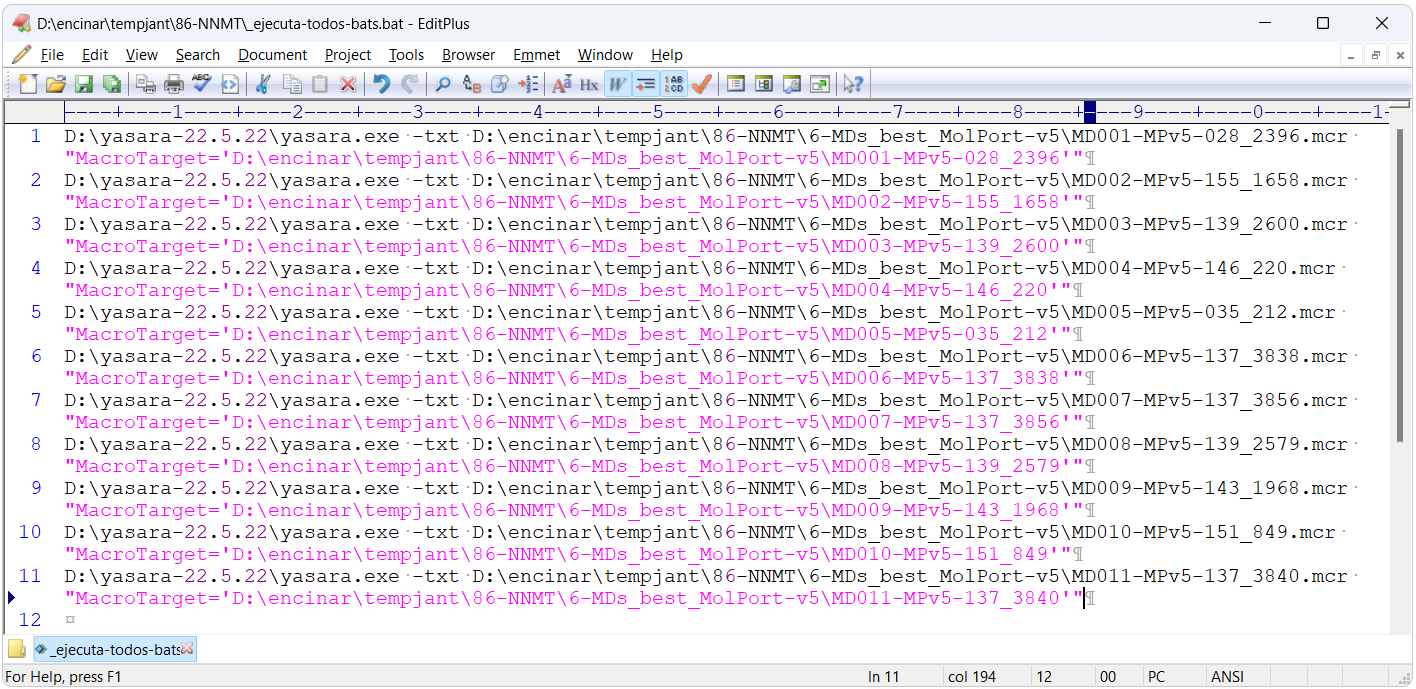
D:\ruta-de-yasara\yasara.exe -txt D:\ruta-del-macro-de-yasara\MD001-MPv5-028_2396.mcr "MacroTarget='D:\ruta-del-macro-de-yasara\MD001-MPv5-028_2396'"Finalmente, el siguiente script de Python, UN-bat-ejecuta-YASARA.py, crea un único fichero _ejecuta-todos-bats.bat (ejecutar en consola en Windows), con tantas líneas como filas tenga en la primera columna el fichero _dos_columnas2.txt, en nuestro ejemplo 11:
...y el fichero _dos_columnas2.txt:
MD001-MPv5-028_2396 MD001-MPv5-028_2396
MD002-MPv5-155_1658 MD002-MPv5-155_1658
MD003-MPv5-139_2600 MD003-MPv5-139_2600
MD004-MPv5-146_220 MD004-MPv5-146_220
MD005-MPv5-035_212 MD005-MPv5-035_212
MD006-MPv5-137_3838 MD006-MPv5-137_3838
MD007-MPv5-137_3856 MD007-MPv5-137_3856
MD008-MPv5-139_2579 MD008-MPv5-139_2579
MD009-MPv5-143_1968 MD009-MPv5-143_1968
MD010-MPv5-151_849 MD010-MPv5-151_849
MD011-MPv5-137_3840 MD011-MPv5-137_3840...y el script PY:
import os
# Rutas de los archivos
template_file = r"D:\6-MDs_best_MolPort-v5\_run-bat.txt"
data_file = r"D:\6-MDs_best_MolPort-v5\_dos_columnas2.txt"
output_file = r"D:\6-MDs_best_MolPort-v5\_ejecuta-todos-bats.bat"
# Leer el contenido del archivo de datos
with open(data_file, 'r') as f:
data_lines = f.readlines()
# Abrir el archivo de salida
with open(output_file, 'w') as output:
# Iterar sobre las líneas de datos
for line in data_lines:
# Dividir la línea en columnas separadas por tabuladores
columns = line.strip().split('\t')
# Leer el contenido de la plantilla
with open(template_file, 'r') as template:
template_content = template.read()
# Reemplazar las variables en la plantilla con los valores de las columnas
modified_content = template_content.replace('[variable-1]', columns[0]).replace('[variable-2]', columns[1])
# Escribir el contenido modificado en el archivo de salida
output.write(modified_content)
output.write('\n')
print("Se ha generado el archivo", output_file)Justo debajo tienes el contenido del nuevo fichero generado: