Cribado viertual de ligandos con Autodock/vina ejecutado sobre YASARA Structure.
Podemos usar YASARA para hacer un cribado de compuestos que se acoplen con baja ΔG (Kcal/mol) a un sitio funcionalmente relevante de una proteína, consiguiendo modular su actividad biológica. Controlaremos el estado de protonación de la proteína y de los ligandos.
Cuando el número de ligandos a testar computacionalmente es elevado, resulta más eficaz ejecutar YASARA en un cluster de computación de alto rendimiento. Aquí usaremos YASARA Structure v20.12.24, un fichero SDF con 176 compuestos en 3D y la estructura 4Y2B.
En https://pubchem.ncbi.nlm.nih.gov/search/search.cgi# generaremos una libreria química con compuestos similares hasta en un 90% al compuesto CCN1C(=O)c2c(c(cs2)c3cccnc3)N=C1NC(C)C, co-cristalizado en 4Y2B.
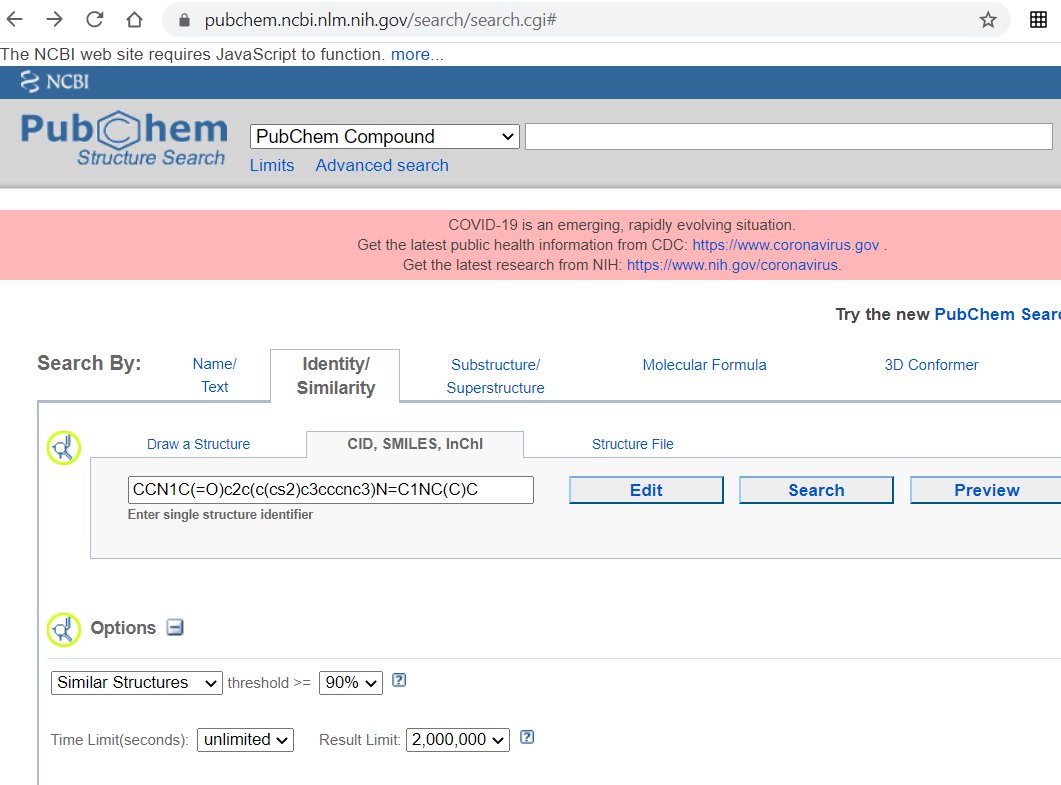
En el momento de es editar este tutorial, encontrados 176 compuestos que cumplan nuestros requisitos de búsqueda.
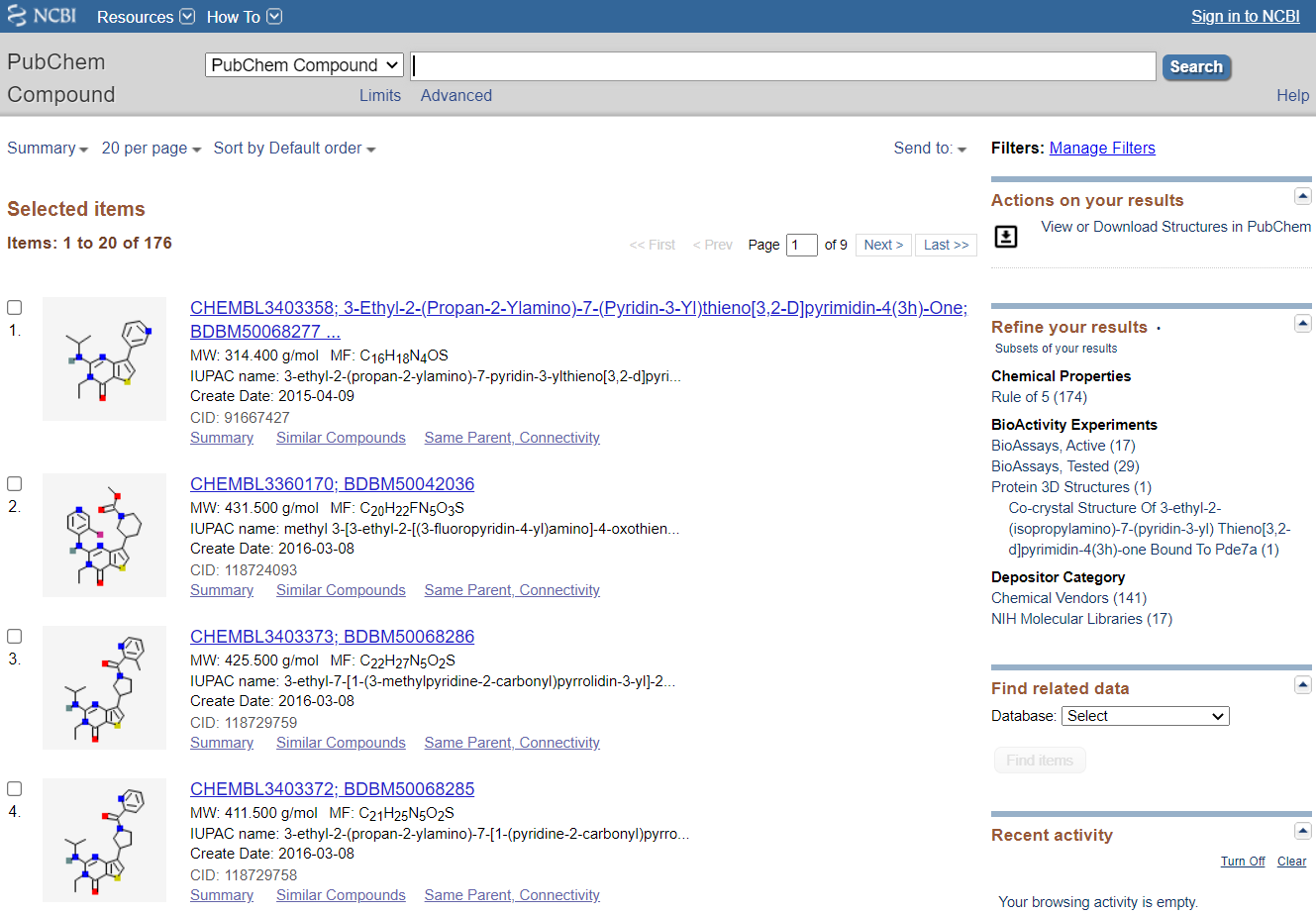
Podemos descargarlos como un fichero SDF en 2D.
Usando DATAWARRIOR v.5.5.0 podemos convertir los compuestos del fichero SDF en 3D. Menú -> Chemistry -> Generate conformers...
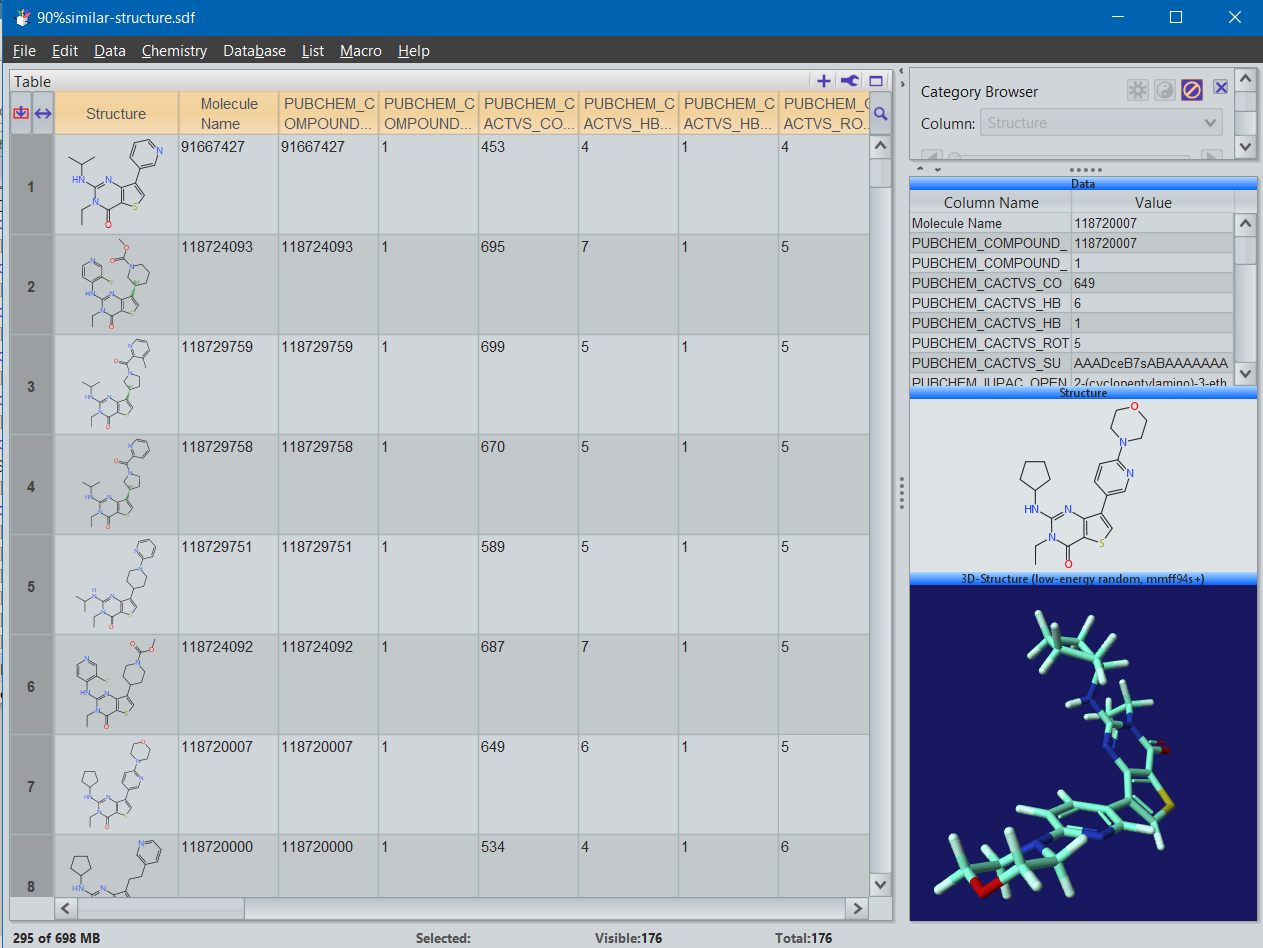
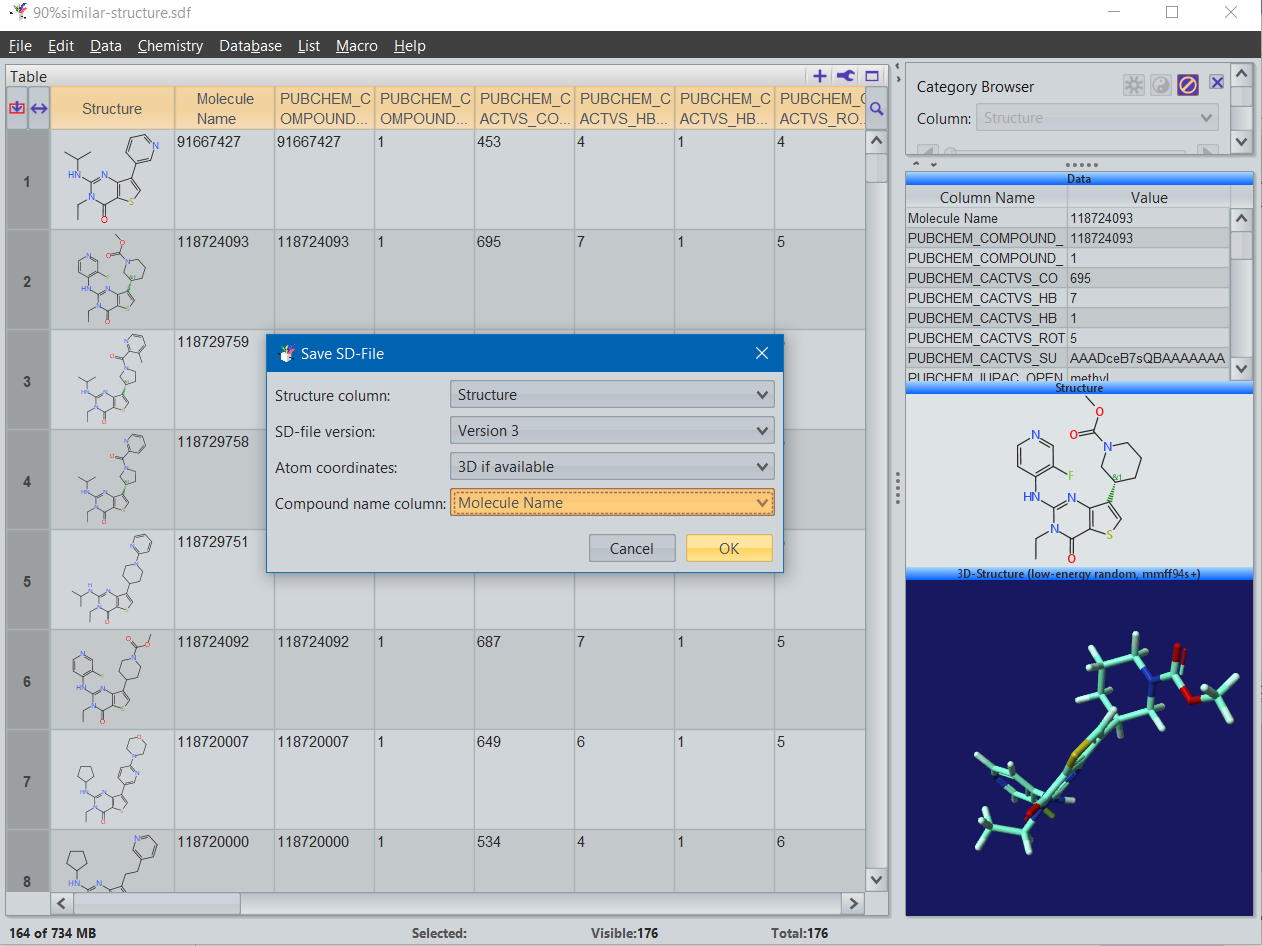
Ahora disponemos de una libreria de 176 compuestos en 3D y vamos a salvarla en formato SDF: Menú -> File -> Save Special -> SDFfile...
Ahora prepararemos una escena de YASARA con la estructura 4Y2B, en la que habremos definido la caja dentro de la cual queremos hacer la simulación de acoplamiento molecular (molecular docking simulation). Empiezo por descargar la estructura 4Y2B usando Pymol, y eliminando las moléculas de agua. Dejamos los átomos de Zn y Mg, junto al inhibidor EPK. Este nos permitirá seleccionar la caja (Grid).
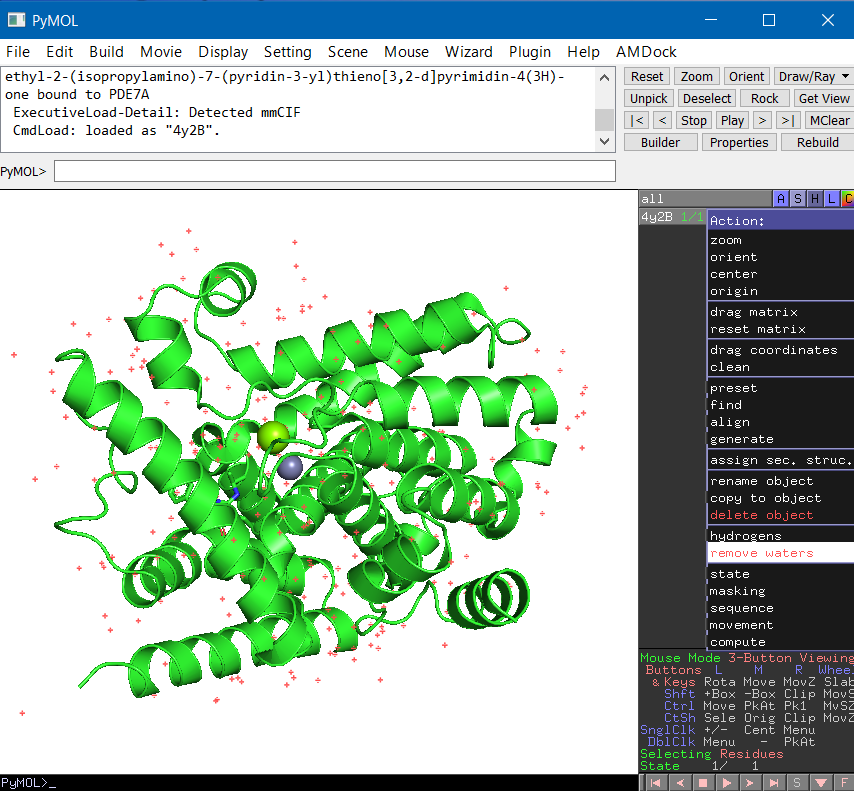
Sobre el margen derecho del escritorio de YASARA desplegamos los objetos, "clic" sobre el Obj 1 -> "clic" derecho -> select -> Newsly -> Epk 583
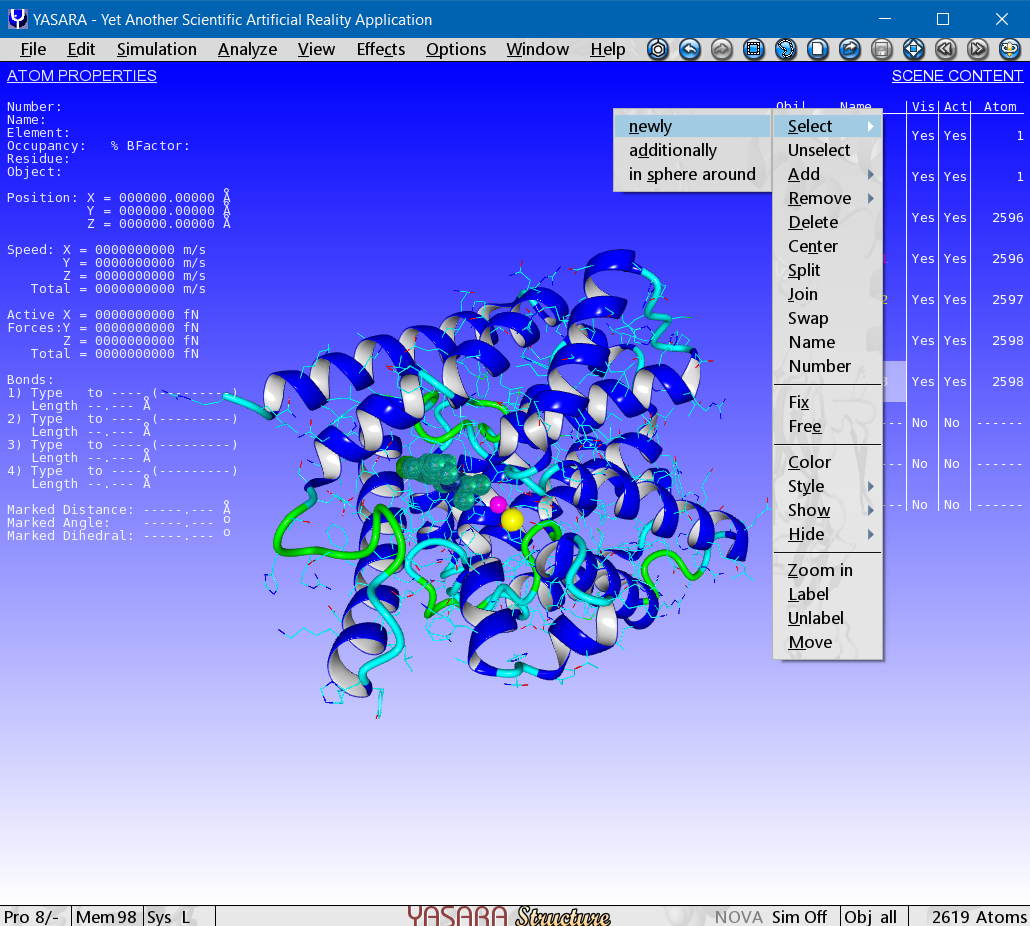
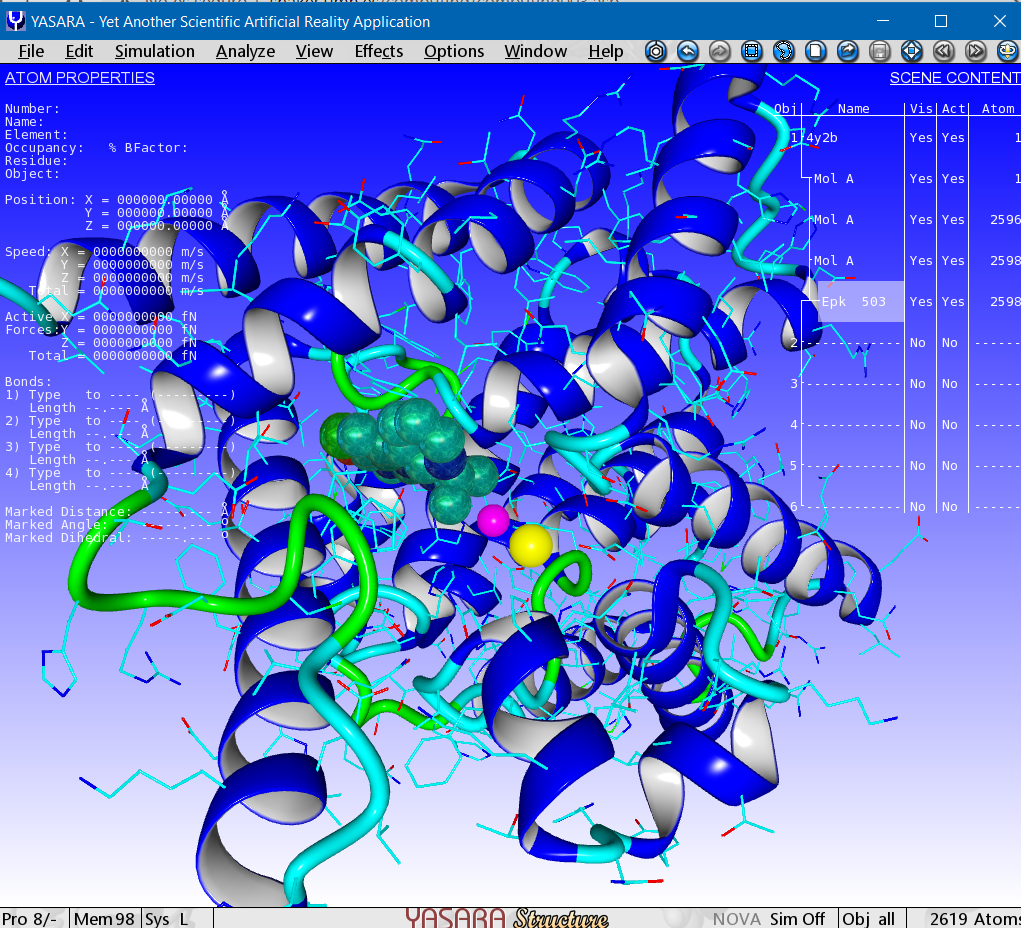
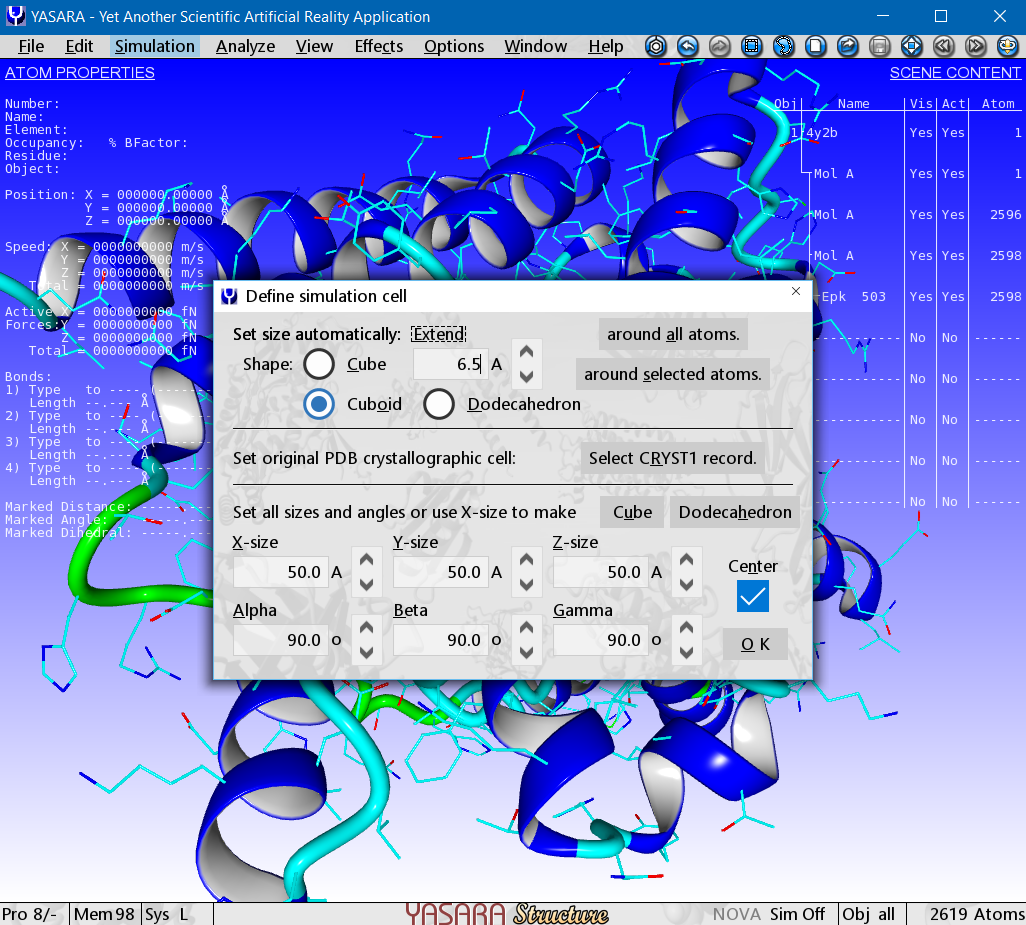
Para seleccionar una caja con centro en el ligando Epk 503 en YASARA: Menú -> Simulation -> Define simulation cell -> Shape: Cuboid -> Extend: 6.5 A -> around selected atoms
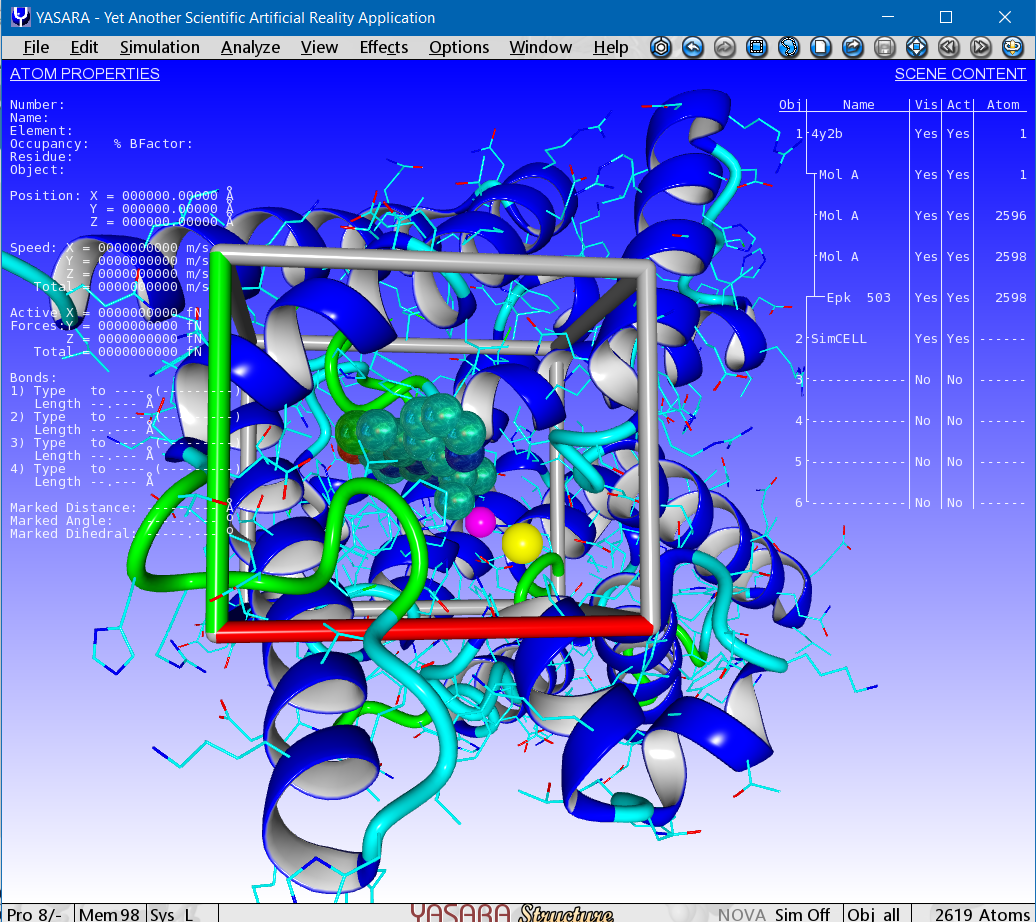
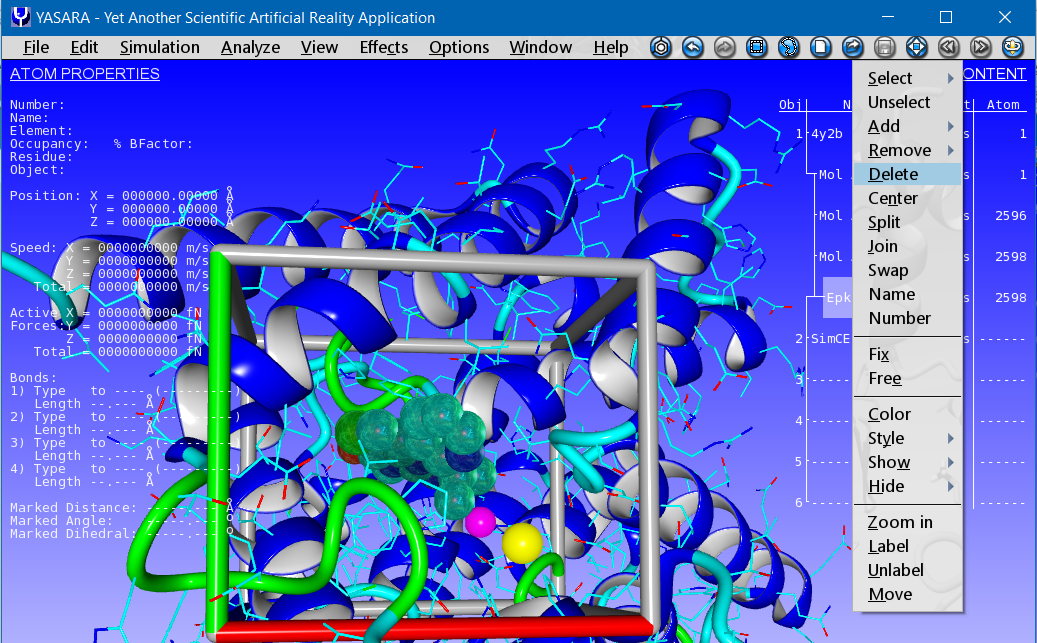
Eliminamos el ligando (Delete) Epk 503 y guardamos una escena de YASARA con el nombre del fichero: 4Y2B_receptor.sce, además, tenemos nuestra libreria de 137 ligandos en 3D, que renombraremos a 4Y2B_ligands.sdf. Esto se describe en Virtual screening of ligands the easy way.
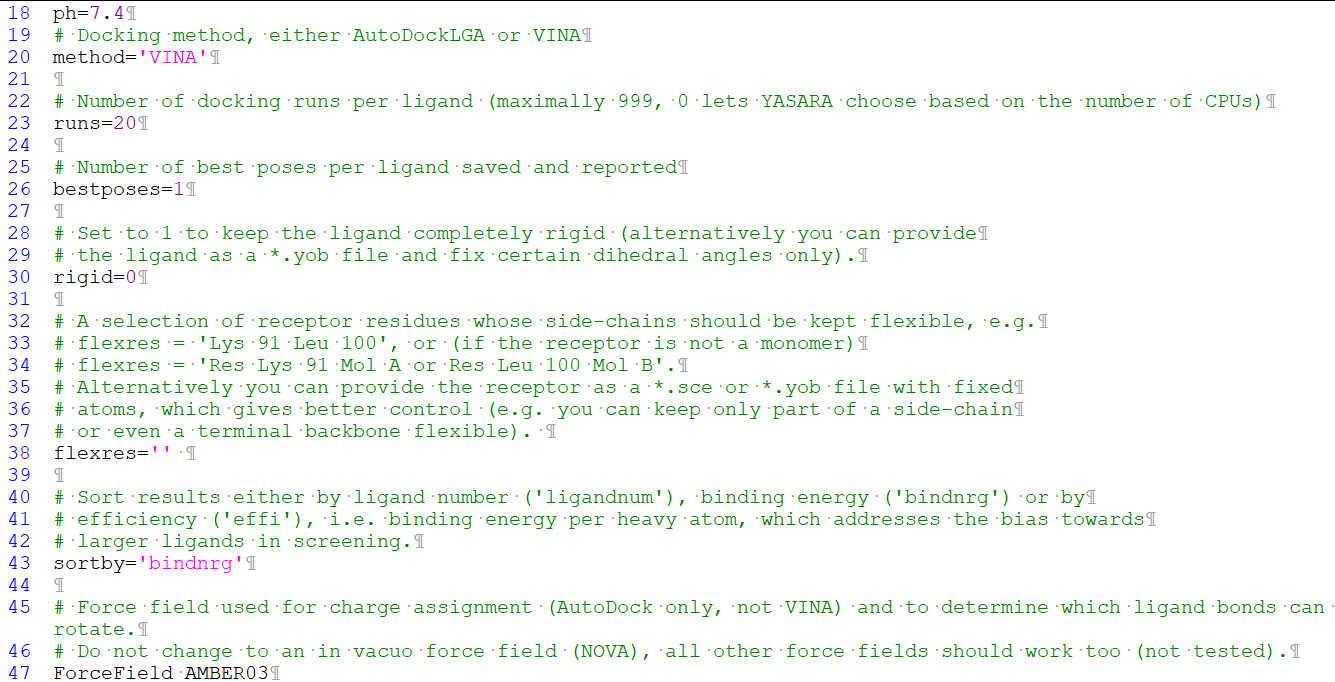
Usaremos un macro de YASARA para hacer el cribado virtual: dock_runscreening.mcr
He editado este macro, añadiendo en la linea 18: ph=7.4, usaré "method='VINA'" (puedo cambiar a "AutoDockLGA'); "runs=20", y podria indicar las cadenas laterales de los aminoácidos situados dentro de la caja que interaccionan con el ligando para que sean flexibles.
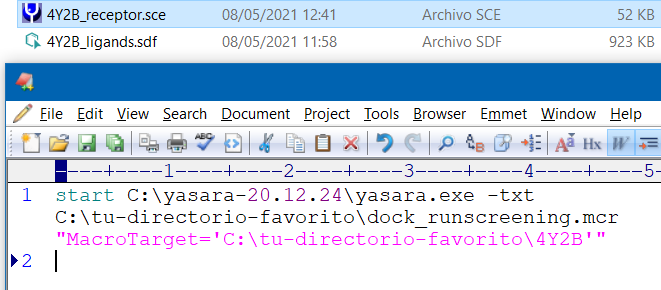
Para ejecutar este macro de YASARA en consola en Windows 10 (¡no en ventana!) edito un fichero _job_run.bat, con el texto que ves justo debajo. Justo doble "clic" sobre él y se inicia el cálculo.
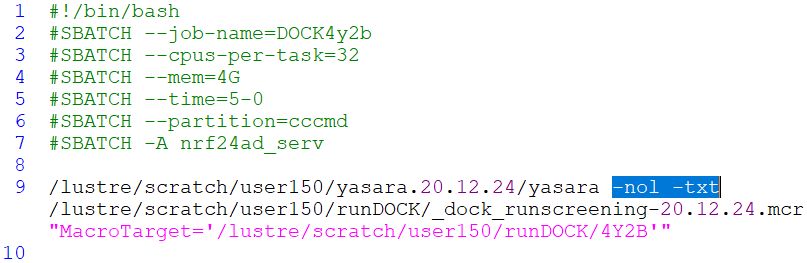
Para ejecutar este macro de YASARA en consola en Linux, con un gestor de colas SLURM, edito un fichero _job_docking.sbatch, con el texto que ves justo debajo.