Preparar ficheros *.pdbqt a partir de ficheros *.pdb (Windows).
Debes tener instalado tanto MGLTools-1.5.2-Setup.exe como python-2.5.2.msi.
Ejecutamos en una línea de comandos (MS-DOS) o en un fichero *.bat una orden similar a la descrita a continuación:
C:\Python25\python.exe "C:\Program Files (x86)\MGLTools 1.5.2\MGLToolsPckgs\AutoDockTools\Utilities24\prepare_receptor4.py" -r D:\aaa\receptor.pdb -o D:\aaa\receptor.pdbqt -A checkhydrogens
C:\Python25\python.exe "C:\Program Files (x86)\MGLTools 1.5.2\MGLToolsPckgs\AutoDockTools\Utilities24\prepare_ligand4.py" -l D:\aaa\ligand.pdb -o D:\aaa\ligand.pdbqt -A checkhydrogensPodemos ejecutar vina_1.2.5_win.exe desde la línea de comandos para calcular ΔG de un complejo receptor-ligando con la siguiente sintaxis:
vina_1.2.5_win.exe --receptor receptor.pdbqt --ligand ligand.pdbqt --size_x 22 --size_y 22 --size_z 22 --autobox --score_only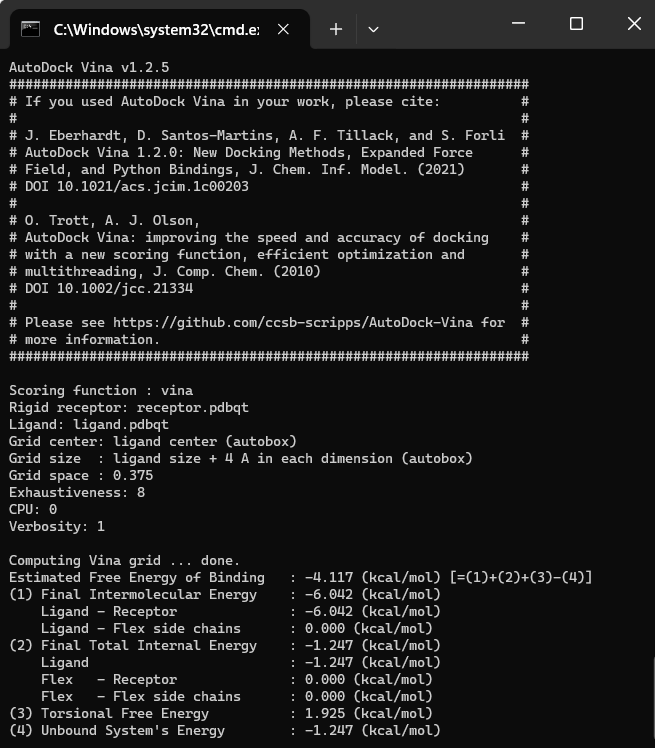
Prepara ficheros *.pdbqt a partir de ficheros *.mol2 (Linux).
1.- Disponemos de una lista de ficheros *.mol2 que queremos convertir en PDBQT. Tenemos en un cluster Linux instalado Marvin 6. Con los siguientes scripts hecemos que todos los nodos del cluster calculen los *.pdbqt lanzando con un solo qsub un array de n jobs (n moléculas).
2.- prepare_ligand4.py se ejecuta con pythonsh.
/home/user/MGLTools-1.5.6rc3/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_receptor4.py -r /home/user/vina/D43A/receptor.POLG_DEN4T-1.pdb -o /home/user/vina/D43A/receptor.POLG_DEN4T-1.pdbqt -A checkhydrogensEn VEGETA el comando es el siguiente...
/share/apps/autodock-4.2.6/mgltools-1.5.6/mgltools_x86_64Linux2_1.5.6/bin/pythonsh /share/apps/autodock-4.2.6/mgltools-1.5.6/mgltools_x86_64Linux2_1.5.6/MGLToolsPckgs/AutoDockTools/Utilities24/prepare_ligand4.py -l /home/jant/chem-library/NITD-2.mol2 -o /home/jant/PDBQT/NITD-1.pdbqt -A checkhydrogens3.- prepare_receptor4.py se ejecuta también con pythonsh.
4.- _genligandstxtVEGETA.py
import sys
import fileinput
import os
diroutconfig='./'
diroutsh='sh_out/'
filelist='_ligands-248143.txt' #sys.argv[1]
fileoutconfig= diroutconfig+'%s.vina_config.txt' #sys.argv[2]
fileoutsh= diroutsh+'%s.sh'
#Create dirs if dont exist
if not os.path.exists(diroutconfig):
os.makedirs(diroutconfig)
if not os.path.exists(diroutsh):
os.makedirs(diroutsh)
templateconfig=open('_template.vina_config-D11B.txt', 'r').read()
templatesh=open('_template.sh.VEGETA.txt', 'r').read()
file = open(filelist, 'r')
for line in file:
comp=line.strip()
print comp
thisfileoutconfig=fileoutconfig %comp.strip()
thisfileoutsh=fileoutsh %comp.strip()
fout=open(thisfileoutconfig, 'w')
fout.write(templateconfig.replace('{{comp}}',comp))
fout.close()
fout=open(thisfileoutsh, 'w')
fout.write(templatesh.replace('{{comp}}',comp))
fout.close()
#break5.- _template.vina_config-D11B.txt
receptor = /home/jant/vina/D11B/receptor.POLG_DEN1B-2.pdbqt
ligand = /home/jant/vina/PDBQT/{{comp}}.pdbqt
center_x = 19.91
center_y = 73.06
center_z = 16.94
size_x = 24.00
size_y = 24.00
size_z = 24.00
out = /home/jant/vina/D11B/{{comp}}.docked.pdbqt
log = /home/jant/vina/D11B/{{comp}}.vina.log
num_modes = 1006.- _template.sh.VEGETA.txt
#!/bin/sh
#
# -- our name ---
#$ -N l-PuCh{{comp}}
#$ -S /bin/sh
#$ -cwd
cd /home/jant/vina/D11B/
/home/jant/vina/autodock_vina_1_1_2_linux_x86/bin/vina --config /home/jant/vina/D11B/{{comp}}.vina_config.txt --log /home/jant/vina/D11B/{{comp}}.log --cpu 2