Medir la ΔG de un ligando cristalográfico con AutoDock/vina.
Prepara un fichero de texto, por ejemplo VS--score_only.csh, e incluye para él propiedades de ejecución con chmod +x VS--score_only.csh
El contenido de este fichero se muestra en la siguiente línea de texto. Incluye la localización de vina, un fichero de configuración: 1AZMa-l.vina_config.txt y el comando --score_only
/home/estudiante/vina/autodock_vina_1_1_2_linux_x86/bin/vina --config 1AZMa-l.vina_config.txt --score_only
vina_1.2.7_win.exe --receptor 1P22-uno.pdbqt --ligand UBJ009.pdbqt --center_x -2.462 --center_y 12.130 --center_z 3.708 --size_x 26.821 --size_y 21.712 --size_z 16.846 --score_only
Puedes calcular la posición del centro del ligando y la caja que cubra al mismo usando un script ya descrito. El contenido de 1AZMa-l.vina_config.txt es el siguiente:
receptor = /home/estudiante/vina/SN002/receptor.1AZMa-r.pdbqt
ligand = /home/estudiante/vina/SN002/1AZMa-l.pdbqt
center_x = 34.27
center_y = 16.43
center_z = -18.71
size_x = 22.00
size_y = 22.00
size_z = 22.00
out = /home/estudiante/vina/SN002/1AZMa-l.docked.pdbqt
log = /home/estudiante/vina/SN002/1AZMa-l.vina.log
num_modes = 100Al ejecutar en consola VS--score_only.csh se generará un fichero de resultados: 1AZMa-l.vina.log cuyo contenido es el siguiente:
#################################################################
# If you used AutoDock Vina in your work, please cite: #
# #
# O. Trott, A. J. Olson, #
# AutoDock Vina: improving the speed and accuracy of docking #
# with a new scoring function, efficient optimization and #
# multithreading, Journal of Computational Chemistry 31 (2010) #
# 455-461 #
# #
# DOI 10.1002/jcc.21334 #
# #
# Please see http://vina.scripps.edu for more information. #
#################################################################
Detected 1 CPU
Reading input ... done.
Setting up the scoring function ... done.
Affinity: -4.29963 (kcal/mol)
Intermolecular contributions to the terms, before weighting:
gauss 1 : 57.12779
gauss 2 : 762.20694
repulsion : 2.39561
hydrophobic : 5.90938
Hydrogen : 1.32201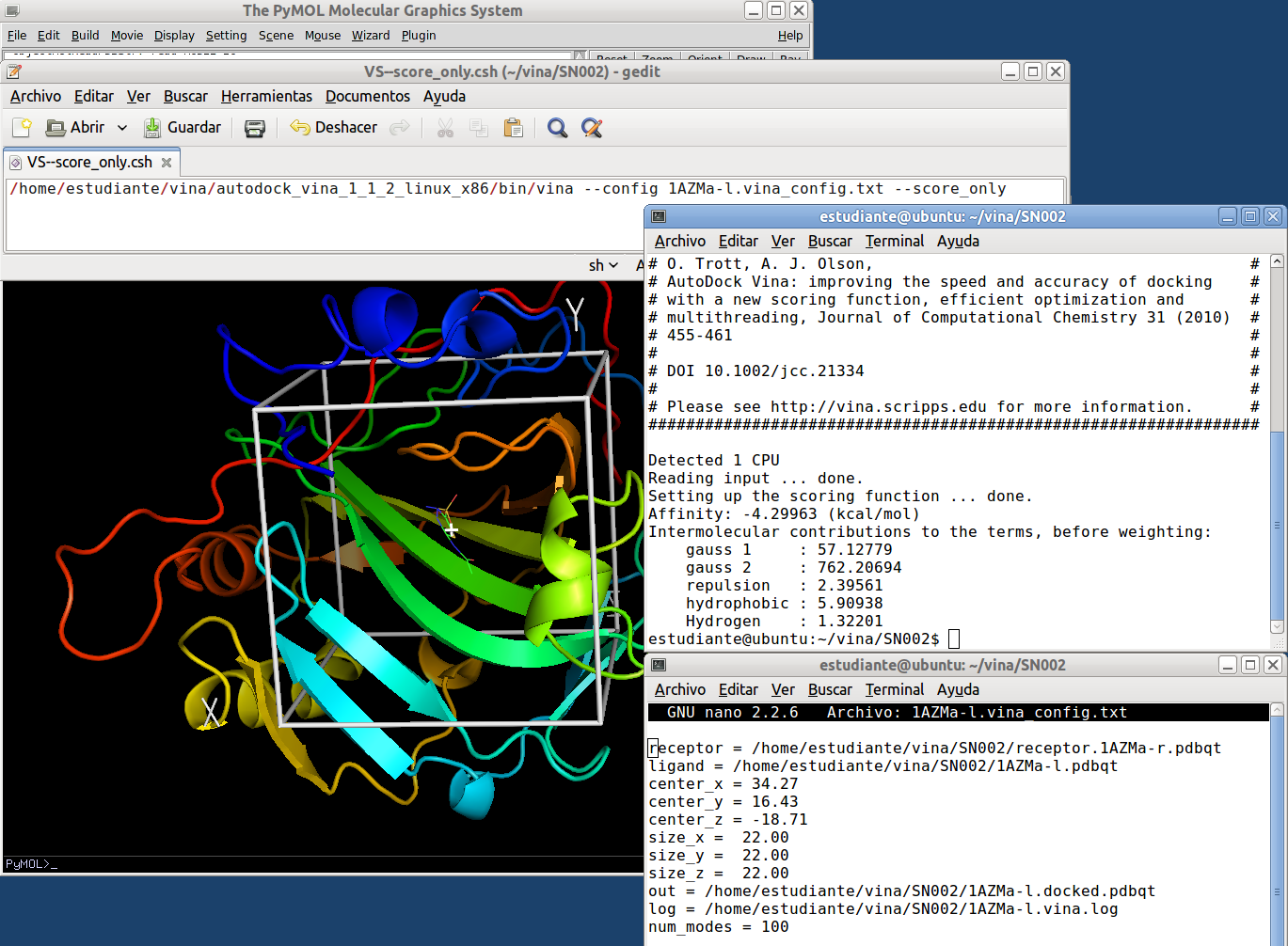
Puedes descargar todos los ficheros en el siguiente enlace.