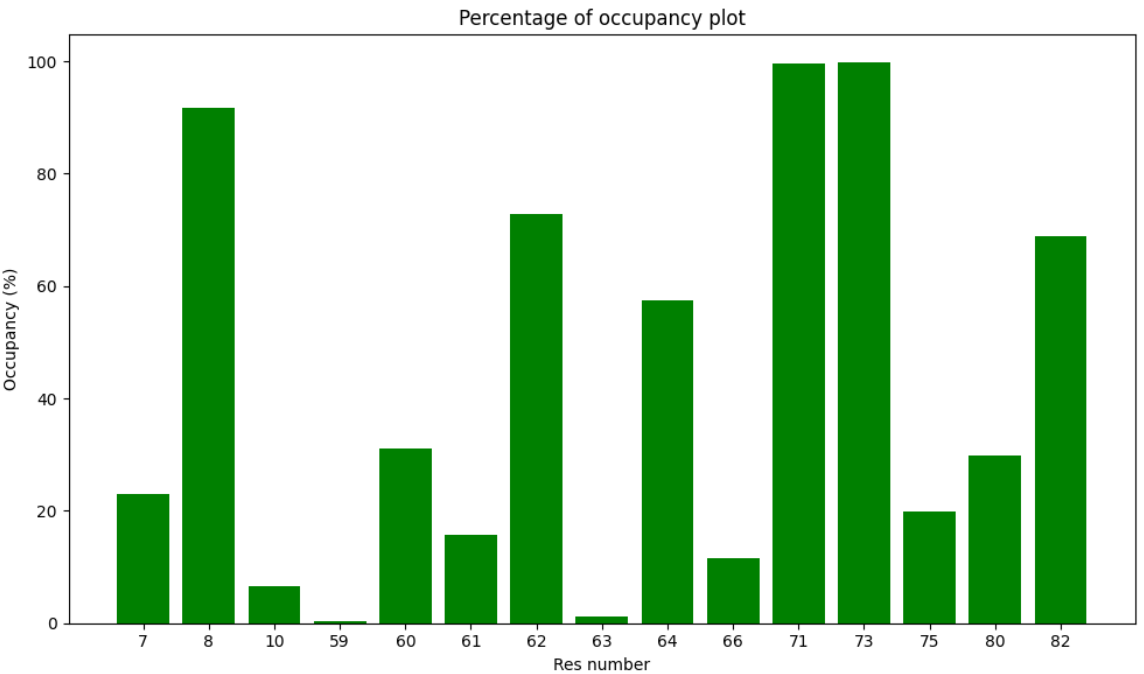
Per-residue ligand interactions of the receptor of molecule A.
El siguiente script de Python permite crear un mapa de color que representa los aminoácidos que están interaccionando con el ligando unido a una proteína durante una simulación de dinámica molecular. Se indica el tipo de interacción. Se requiere una matriz de datos separados por tabuladores como la que se usa en este ejemplo.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# Función para asignar colores según el valor de la celda
def get_color(val):
color_map = {
1: 'white',
2: 'red',
3: 'green',
4: 'yellow',
5: 'blue',
6: 'magenta',
7: 'cyan',
8: 'gray'
}
return color_map.get(val, 'white') # 'white' como valor por defecto si no está en el mapa
# Leer el archivo delimitado por tabuladores
file_path = r'E:\aaa\52-UNL1-A.txt'
# Usamos pandas para leer el archivo
df = pd.read_csv(file_path, delimiter='\t')
# Extraer la primera columna (Simulation time in nanoseconds) para el eje X
x_labels = df.iloc[:, 0].values
# Extraer el resto de las columnas (a partir de la segunda) para los datos
data = df.iloc[:, 1:].to_numpy()
# Crear la figura
fig, ax = plt.subplots(figsize=(15, 6))
# Generar una matriz de colores en función de los valores de la matriz
colors_matrix = np.vectorize(get_color)(data)
# Dibujar la matriz de colores manualmente con un bucle
for (i, j), val in np.ndenumerate(data):
ax.add_patch(plt.Rectangle((i, j), 1, 1, color=get_color(val)))
# Ajustar los límites de los ejes para que coincidan con el tamaño de los datos
ax.set_xlim(0, data.shape[0])
ax.set_ylim(0, data.shape[1])
# Etiquetas y título
ax.set_title('Per-residue ligand interactions of the receptor of molecule A')
ax.set_xlabel('Simulation time in nanoseconds')
ax.set_ylabel('Res number')
# Ajustar las etiquetas del eje X para que se muestren cada 10 nanosegundos
tick_spacing = 10 # Cada 10 nanosegundos
x_range = np.arange(0, 201, tick_spacing) # Generar 21 marcas, desde 0 hasta 200 inclusive
# Aplicar las posiciones de las marcas de graduación en el eje X
ax.set_xticks(np.linspace(0, data.shape[0] - 1, len(x_range))) # Alineación de las marcas con los datos
# Establecer las etiquetas correspondientes en el eje X (cada 10 nanosegundos)
ax.set_xticklabels(x_range)
# Definir las etiquetas de la leyenda
legend_labels = {
1: 'None',
2: 'HB',
3: 'Hyd',
4: 'Hyd+HB',
5: 'Ion',
6: 'Ion+HB',
7: 'Ion+Hyd',
8: 'Ion+Hyd+HB'
}
# Crear leyenda
handles = [plt.Line2D([0], [0], marker='s', color='w', markerfacecolor=get_color(i), markersize=10) for i in range(1, 9)]
ax.legend(handles, [legend_labels[i] for i in range(1, 9)], loc='upper right', bbox_to_anchor=(1.3, 1))
# Mostrar la gráfica
plt.tight_layout()
plt.show()El gráfico generado es el siguiente:
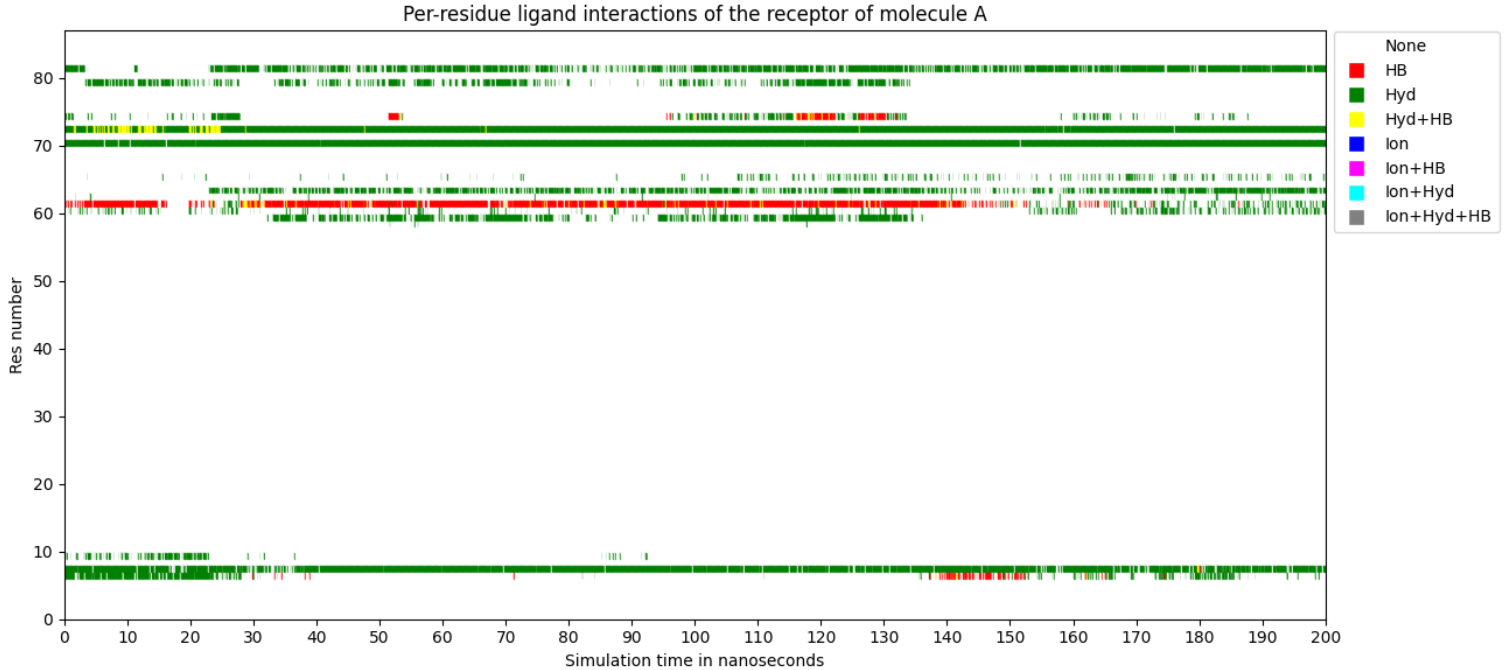
El siguiente script de Python para ejecutar en Windows permite calcular el porcentage de ocupación de cada aminoácido que interacciona con el ligando durante la simulación de dinámica molecular. Usa la misma matriz de datos que el script anterior.
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
# Leer el archivo delimitado por tabuladores
file_path = r'E:\aaa\52-UNL1-A.txt'
# Usamos pandas para leer el archivo
df = pd.read_csv(file_path, delimiter='\t')
# Extraer la primera columna (Simulation time in nanoseconds) para el eje X
x_labels = df.iloc[:, 0].values
# Extraer el resto de las columnas (a partir de la segunda) para los datos
data = df.iloc[:, 1:].to_numpy()
# Filtrar valores mayores que 1 y calcular porcentaje para cada "Res number" (es decir, cada columna)
percentages = np.mean(data > 1, axis=0) * 100 # Calcular porcentaje de valores > 1 para cada columna
# Filtrar los índices donde el porcentaje es mayor que 0
non_zero_indices = np.where(percentages > 0)[0]
filtered_percentages = percentages[non_zero_indices]
filtered_res_numbers = non_zero_indices + 1 # Ajustar índices para que correspondan a los valores de "Res number"
# Mostrar la tabla de valores
print("Tabla de valores con porcentage de ocupación de cada aminoácido:")
print(f"{'Residue number':<12}{'Percentage, (%)':<20}")
for res, pct in zip(filtered_res_numbers, filtered_percentages):
print(f"{res:<12}{pct:<20.2f}")
# Crear gráfico de barras con etiquetas categóricas en el eje X
fig, ax = plt.subplots(figsize=(10, 6))
# Dibujar las barras
ax.bar(filtered_res_numbers.astype(str), filtered_percentages, color='green') # Convertimos Res numbers a string para usar como etiquetas categóricas
# Etiquetas y título
ax.set_title('Percentage of occupancy plot')
ax.set_xlabel('Res number')
ax.set_ylabel('Occupancy (%)')
# Mostrar gráfico
plt.tight_layout()
plt.show()Tabla de valores con porcentage de ocupación de cada aminoácido:
Residue number Percentage, (%)
7 23.04
8 91.80
10 6.50
59 0.30
60 31.18
61 15.69
62 72.81
63 1.15
64 57.52
66 11.64
71 99.60
73 99.75
75 19.79
80 29.84
82 68.92