miniconda, RDkit, vina, tkdm, OpenBabel, Autodock-vina y estereoisómeros R/S.
¿Podemos instalar en nuestro /home de Linux miniconda sin ser administrador?
Sí, podemos instalar Miniconda en nuestro directorio /home en Linux sin necesidad de privilegios de administrador. Esta es una de las ventajas principales de Miniconda (y Anaconda): puede instalarse en modo "user-local", es decir, en tu espacio personal sin afectar al sistema global.
Descarga el instalador desde el sitio oficial:
wget https://repo.anaconda.com/miniconda/Miniconda3-latest-Linux-x86_64.sh -O ~/miniconda.shDale permisos de ejecución al script:
chmod +x ~/miniconda.shEjecuta el instalador:
./miniconda.shDurante la instalación: Elige un directorio dentro de tu /home, por ejemplo: /home/tu_usuario/miniconda3. Y di "yes" cuando te pregunte si deseas inicializar conda en tu .bashrc o .zshrc.
Activa la instalación ejecutando:
source ~/miniconda3/etc/profile.d/conda.sh
conda initA partir de ahí podrás usar:
conda create -n mi_entorno python=3.10
conda activate mi_entornoVerifica que conda está instalado.
conda infoCrear el entorno virtual llamado RD-openbabel
conda create -n RD-openbabel
conda activate RD-openbabel # para activar el entorno recién creado
conda install -c conda-forge openbabel # instalar openbabel o algún otro paquete,
# hazlo mientras el entorno esté activado
obabel -V # verificar la versión de openbabel
Open Babel 3.1.1 (o versiones más recientes) no es compatible con Python 3.6.8. A partir de Open Babel 3.1.0, se requiere al menos Python 3.7 para instalar y utilizar la biblioteca.
¿Cómo instalo RDkit, tqdm y Autodock vina 1.2.5?
conda install -c conda-forge rdkit
conda install -c conda-forge tqdm
conda install -c conda-forge vina=1.2.5
# Después de instalar, puedes verificar que todo funciona correctamente:
python -c "import rdkit; print(rdkit.__version__)"
python -c "import tqdm; print(tqdm.__version__)"
vina --version
# Para verificar que todos los paquetes están instalados correctamente en el entorno:
conda listGeneración de estereoisómeros R/S y E/Z.
Script de Python para ejecutar en Linux que genera estereoisómeros R/S y E/Z, partiendo de un fichero de texto separado en por tabuladores que tenga en la primera columna el nombre de la molecula (sin tildes, ni caracteres latinos, ni espacios) y en la segunda columna el SMILES.
import os
from rdkit import Chem
from rdkit.Chem import AllChem
from rdkit.Chem.EnumerateStereoisomers import EnumerateStereoisomers, StereoEnumerationOptions
import subprocess
# Input and output paths
input_file = "/home/jant.encinar/py-linux/39_smiles-a-SDF3DyH/_name-smiles.txt"
output_file = "/home/jant.encinar/py-linux/39_smiles-a-SDF3DyH/_SDF-salida-3D-H.sdf"
temp_sdf = "/home/jant.encinar/py-linux/39_smiles-a-SDF3DyH/temp_isomer.sdf"
babel_output = "/home/jant.encinar/py-linux/39_smiles-a-SDF3DyH/temp_out.sdf"
# Enumerate all stereoisomers including assigned stereocenters (R/S) and double bonds (E/Z)
options = StereoEnumerationOptions(
tryEmbedding=True,
onlyUnassigned=False, # Enumerate all stereoisomers, assigned and unassigned
unique=True,
maxIsomers=128
)
with open(output_file, 'w') as out_global:
with open(input_file, 'r') as f:
for line in f:
line = line.strip()
if not line or '\t' not in line:
continue
name, smiles = line.split('\t')[:2]
mol = Chem.MolFromSmiles(smiles)
if mol is None:
print(f"Failed to parse SMILES for {name}: {smiles}")
continue
isomers = list(EnumerateStereoisomers(mol, options=options))
for idx, isomer in enumerate(isomers):
isomer = Chem.AddHs(isomer)
# Generate 3D coordinates
if AllChem.EmbedMolecule(isomer, AllChem.ETKDG()) != 0:
print(f"3D embedding failed for {name}_{idx+1}")
continue
AllChem.UFFOptimizeMolecule(isomer)
# Set molecule name with suffix if multiple isomers
isomer_name = f"{name}_{idx+1}" if len(isomers) > 1 else name
isomer.SetProp("_Name", isomer_name)
Chem.MolToMolFile(isomer, temp_sdf)
# Run Open Babel to add hydrogens and refine 3D geometry
command = [
"obabel", temp_sdf, "-O", babel_output,
"-h", "--gen3d"
]
result = subprocess.run(command, stderr=subprocess.PIPE, text=True)
if result.returncode != 0 or not os.path.exists(babel_output):
print(f"Open Babel error for {isomer_name}: {result.stderr}")
continue
with open(babel_output, 'r') as babel_file:
content = babel_file.read()
out_global.write(content)
print("All done. Output written to:", output_file)
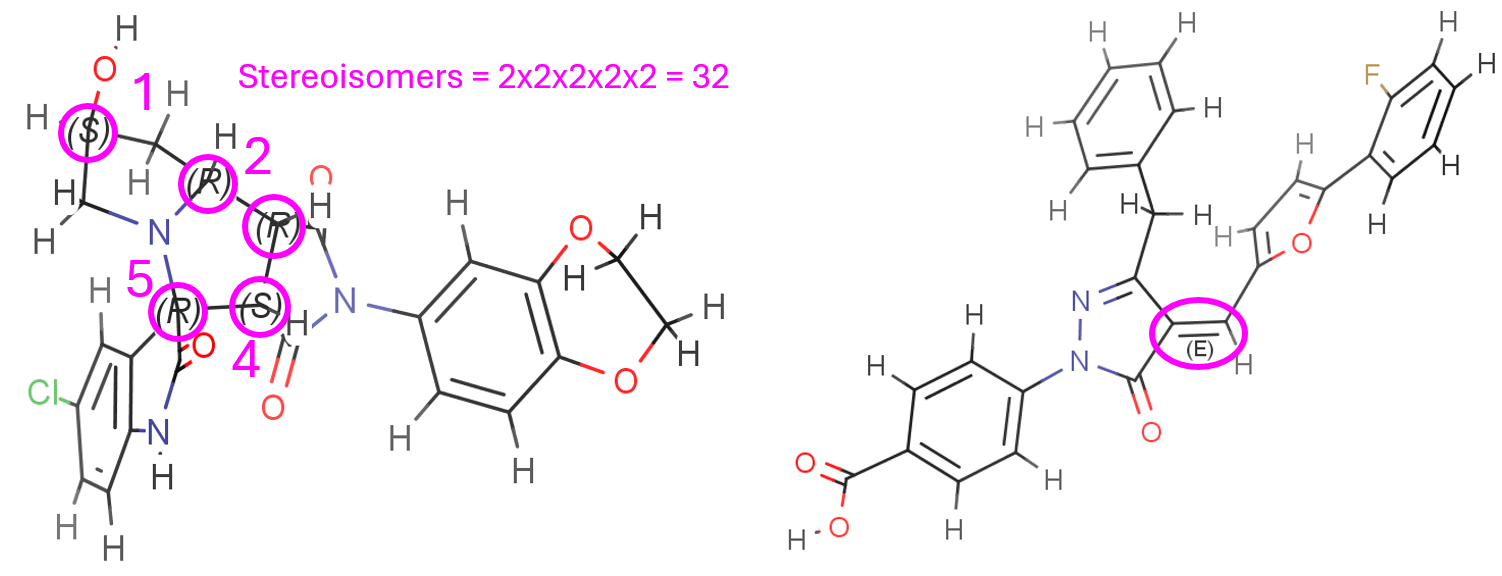
Fichero de entrada: _name-smiles.txt
Fichero de salida: _SDF-salida-3D-H.sdf. La primera molécula tiene 5 carbonos quirales, que permitirian pensar en 32 estereoisómeros diferentes. El script los ha generado. La segunda molécula de entrada tiene un doble enlace, el script ha generado tanto el isómero CIS como el TRANS.