ANÁLISIS MATEMÁTICO DETALLADO DEL CÁLCULO DEL COEFICIENTE DE PARTICIÓN EN LOS EXPERIMENTOS DE UNIÓN DE PEPTIDO-NBD A VESICULAS LIPÍDICAS.
Bibliografía: Gazit, E. y Shai, Y. (1993). Biochemistry. 32: 12363-12371.
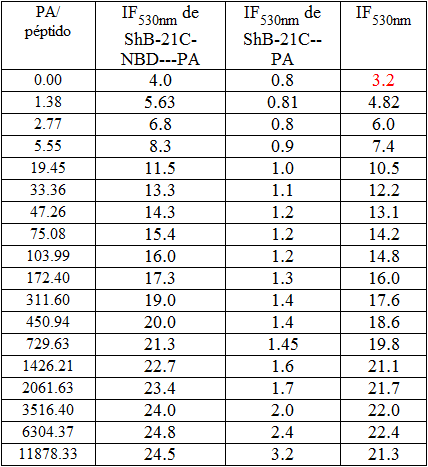
Se recoge como ejemplo los datos reales de uno de los experimentos de unión del péptido ShB-21C-NBD a vesículas unilamelares pequeñas de PA.
A alícuotas de péptido ShB-21C-NBD de concentración 0.1 mM se le añaden vesículas unilamelares pequeñas (SUV) de PA para conseguir distintas relaciones lípido-péptido y se mide la intensidad de fluorescencia inmediatamente después de la mezcla. La longitud de onda de excitación es de 470 nm y la de emisión de 530 nm. Se corrige la fluorescencia de la mezcla ShB-21C-NBD/PA restando la dispersión de la luz producida por la mezcla ShB-21C/PA. Estos datos iniciales se recogen en la Tabla 1.
Tabla 1: intensidades de fluorescencia vs relación molar lípido/péptido.
La representación de estos datos se recoge en la Fig. 1:
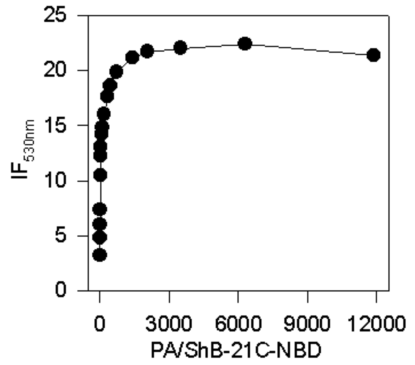
Fig. 1: Curva de unión del péptido ShB-21C-NBD a SUV de PA
Las constantes de unión se analizan como un equilibrio de partición de acuerdo con la ecuación:
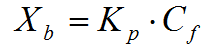
donde Xb se define como la relación molar del péptido unido (Cb) por lípido total (CL), Kp es el coeficiente de partición, y Cf representa la concentración de péptido libre (no unido) en el equilibrio. Para calcular Xb es necesario conocer primero fb (es decir, la fracción del péptido unido a membrana), que se calcula utilizando la ecuación:
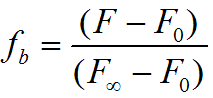
en la que F∞ (la señal de fluorescencia que se obtendría si todo el péptido se une al lípido), que podría calcularse ajustando los datos los datos de la Fig. 1 a una ecuación hiperbólica similar a la que explica la cinética michaeliana o extrapolando a partir de la representación doble inversa de F (intensidad de la fluorescencia observada, es decir, IF530nm recogido en la Tabla 1) y de la CL (concentración total de lípido); equivalente a la representación de Lineweaver-Burk.
Tabla 2: Datos para la representación de dobles inversos.
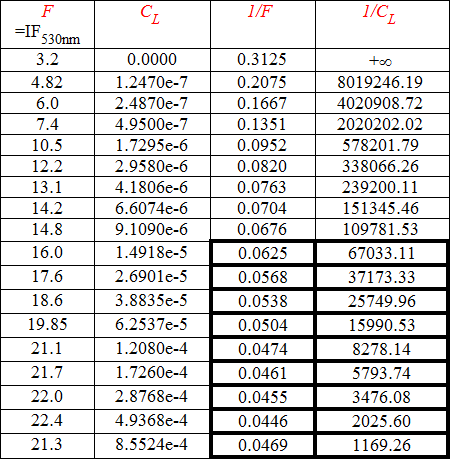
Teniendo que para los valores pequeños de CL, pequeños errores de F conducen a grandes errores de 1/F, lo que supondría grandes errores de F∞, despreciamos los valores más pequeños de la Tabla 2. El valor de la ordenada en el origen de la ecuación de la recta a la que se ajustan estos puntos será el inverso de F∞.
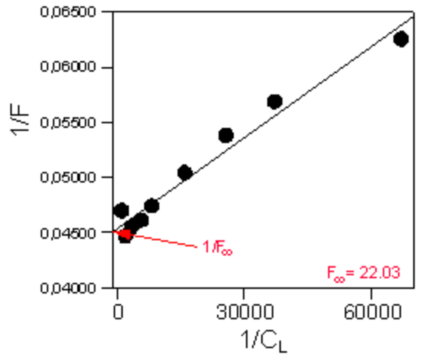
Fig. 2: Representación de dobles inversos para calcular F∞
Conociendo F∞ podremos calcular la fracción del péptido unido a membrana (fb):
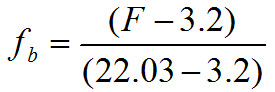
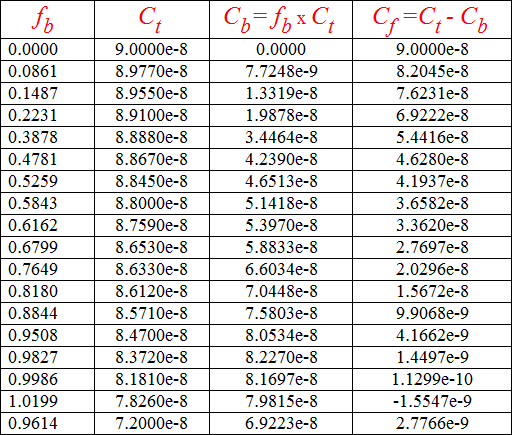
Los datos obtenidos para fb aparecen en la Tabla 3. En ella también aparecen los datos de concentración de péptido total (Ct) (corregidos para el volumen ligeramente distinto en cada relación lípido-péptido). Multiplicando fb por la concentración de péptido total obtenemos la concentración de péptido unido (Cb). La concentración de péptido libre (Cf) = (Ct)-(Cb). Asumiendo que los péptidos se unen sólo a la hemicapa externa de los liposomas (60% del lípido total) debemos corregir el valor de Xb como:
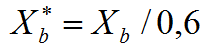
por lo que la ecuación de partición se convierte en:
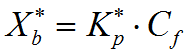
En la Tabla 3 se recogen todos estos parámetros para el ejemplo elegido:
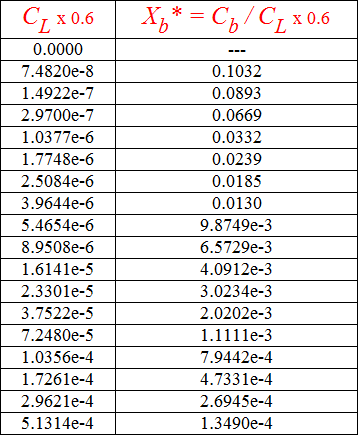
La representación Xb* versus Cf es lo que comunmente se conoce como isoterma de unión, y que queda representado en la Fig. 3.
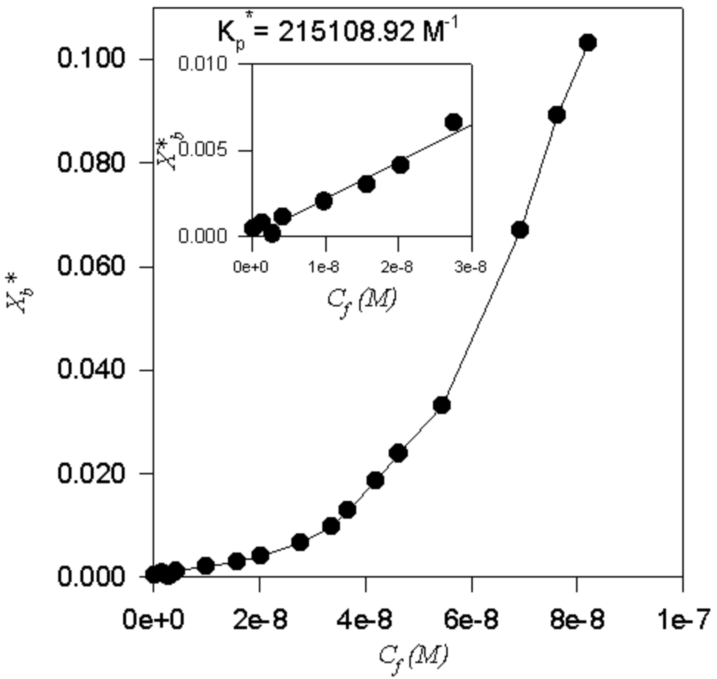
El tramo inicial de la isoterma de unión se puede ajustar a una recta que pase por el origen, coincide precisamente con el tramo final o “plato” de la curva de unión (Fig. 1). La pendiente de esta recta será Kp* que para el ejemplo tomado tiene un valor de 249.674, 03 M-1.